关键是基于shapely模块中的within方法,判断信号点与ROI的包含关系。
### 根据单细胞分割的ROI进行过滤
from roifile import roiread
from shapely import geometry as geo
from shapely import within
fp = r"D:\data\xxd\250219\s2-z5-1_rois.zip"
# cellpose 分割后导出的roi记录,ImageJ兼容
rois = roiread(fp)
pts = rois[0].integer_coordinates
pixelsize = 0.0313
# 单位与 imaris 保持一致,为微米
roi = geo.Polygon(pts*pixelsize)
info = []
for idx, row in data2.iterrows():
p = geo.Point([row['x'], row['y']])
if within(p, roi):
info.append([idx, row['mean'], row['volume'], row['Nearest 1 Neighbors Distance'], 1])
# idx 为 cluster 的索引,最后一个为细胞的索引
data3 = pd.DataFrame(data=info, columns=['pid', 'mean', 'volume', 'NND', 'cell'])注意 ROI 记录是 cellpose 分割出来的。
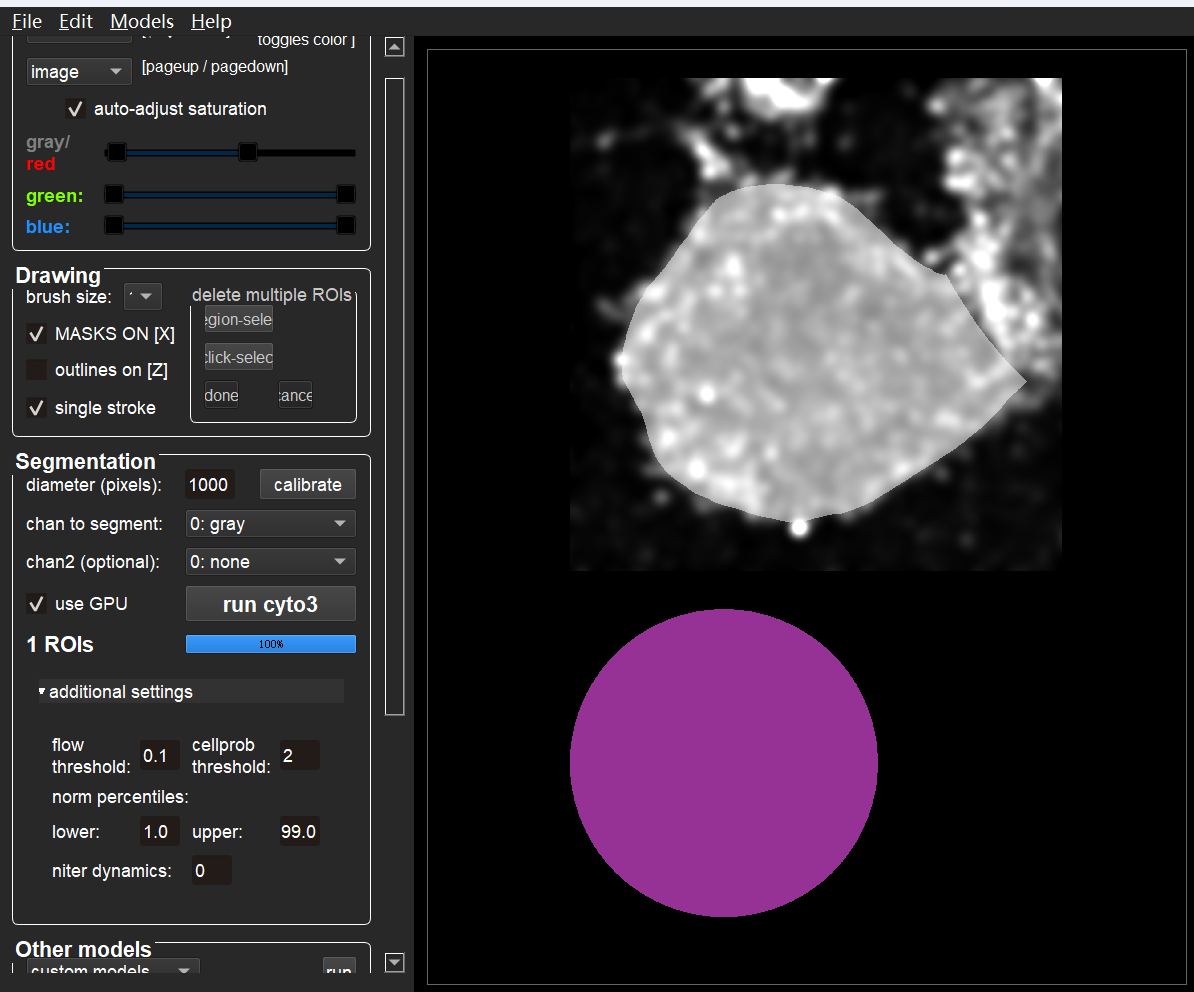
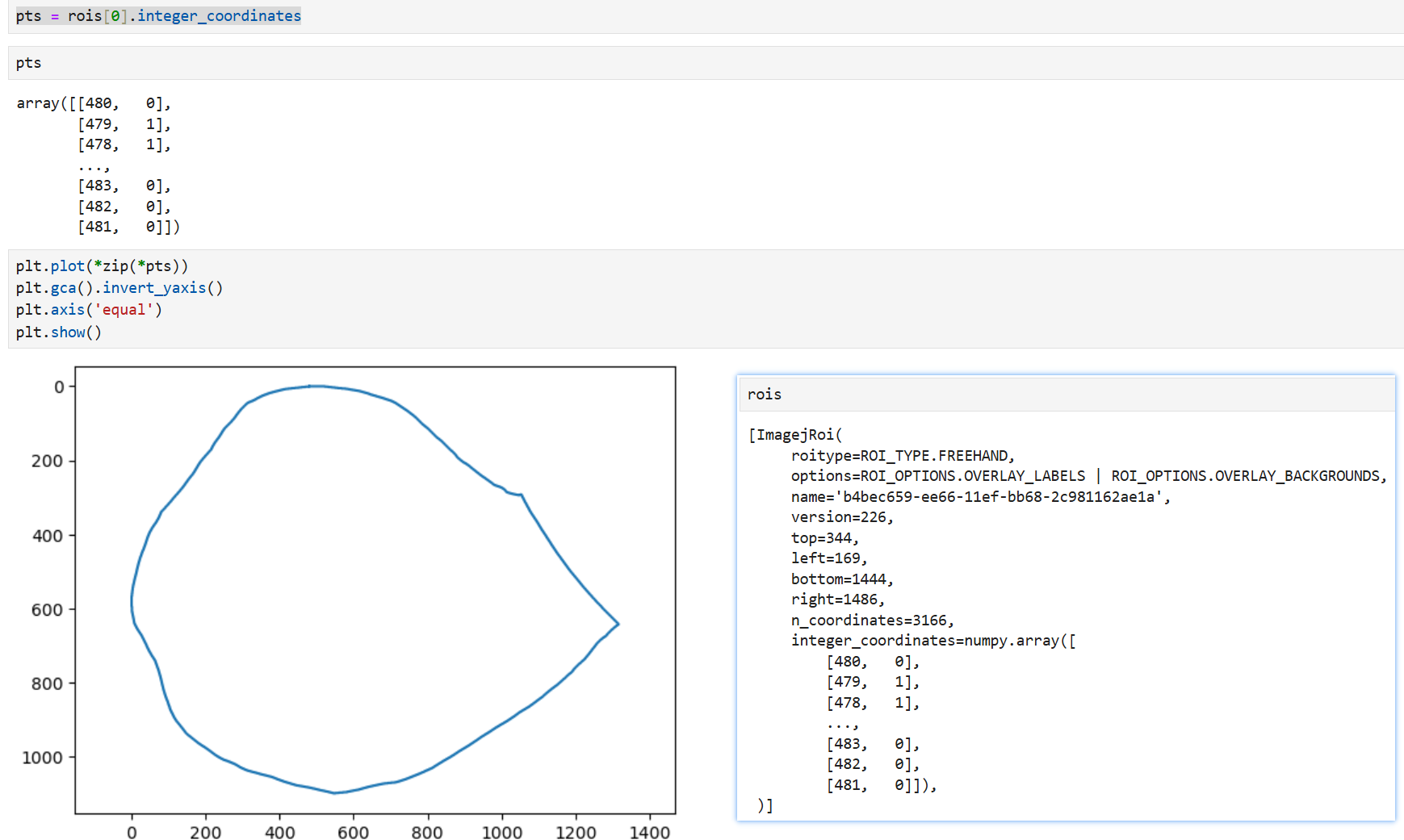
python中可以使用 roifile 模块读取,顺带检查一下,是对的。

此处评论已关闭