使用编程的方式能够高度自定义地对大量数据进行分析,但为保证分析的准确性,需要对中间步骤进行充分的可视化检查。
由于实际采集的数据通常比较“脏”(即存在你没有考虑到的情况),所以如果基于少数抽样样本来设计算法并执行分析程序,大概率会得到错误的结果。如果分析结果奇怪还好,这会倒逼你回过头去逐步检查。最怕的是直接得出一个符合预期的结果。这里记录一个分析信噪比数据的案例。
from roifile import roiread
from aicsimageio import AICSImage
from skimage.draw import polygon
import os
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
def getFileList(wks, ext):
'''ext 为文件后缀名'''
flist = []
for root, ds, fs in os.walk(wks):
for fname in fs:
if fname.endswith(ext):
fpath = os.path.join(root, fname)
flist.append(fpath)
return flist
def get_values(img, roi):
'''根据roi从图像中截取矩形范围'''
coords = roi.coordinates()
c = coords[:, 0]
r = coords[:, 1]
rows, cols = polygon(r, c)
values = img[rows, cols].flatten()
return values
def calculateSNR(a:list, b:list)->dict:
'''
a: signal+background intensity
b: background intensity
'''
sig_bk = np.mean(a)
bk = np.mean(b)
sig = sig_bk - bk
sig_bk_std = np.std(a)
bk_std = np.std(b)
sig_std = np.sqrt(pow(sig_bk_std, 2)+pow(bk_std, 2))
SNR = sig/bk
SNR_std = SNR * np.sqrt(pow(sig_std/sig, 2) + pow(bk_std/bk, 2))
res = {
"MEAN_SIG+BACK": sig_bk,
"SD_SIG+BACK": sig_bk_std,
"MEAN_BACK": bk,
"SD_BACK": bk_std,
"MEAN_SIG": sig,
"SD_SIG": sig_std,
"SNR": SNR,
"SD_SNR": SNR_std,
}
return res上述代码就包含了计算了信噪比的关键函数 calculateSNR,该方法来源于文献,需要同时输入信号的像素值(相机采集的信号区域,通常包含了背景),以及纯背景区域的像素值。然后采取均值相减的方式(sig_bk - bk = sig)获得实际的信号强度 sig,然后除以噪声均值 bk 得到信噪比 SNR。需要注意的是该方法默认噪声和信号的像素值都服从正态分布,所以选取ROI对信号和噪声的像素值进行采样时,注意避免人为偏误。

理论上上述代码能够完成SNR分析,但用户希望利用这个方法来实现细胞膜区域的一种特殊SNR分析,信号是他的方法增强的,而背景则是未使用该方法的对照组细胞膜区域的背景信号强度(存在细胞自发荧光的考虑)。这种基于指定object region从不同图像中采集sig和bk 的SNR分析也是可行的,出于描述简便,这种SNR分析本文简称为 cross-image SNR,然而因为数据量比较大,数据一致性就难以保证,所以就不得不进行必要的可视化。
fps = getFileList(wks='./', ext='czi')
df = {}
for idx, fp in enumerate(fps):
n_channel = fp[2] # 多少色
pp = fp.split("\\")
cell = pp[1] # 细胞类型
etype = pp[2] # 对照或实验组
rid = idx%2 + 1 # replicate次数
fp2 = fp.replace(".czi", ".rois.zip")
rois = roiread(fp2)
img = AICSImage(fp)
pixelsize = img.physical_pixel_sizes.X
channels = img.channel_names
for idy, ch in enumerate(channels):
ch_ = channel_alt[ch]
if n_channel not in df:
df[n_channel] = {}
if cell not in df[n_channel]:
df[n_channel][cell] = {}
if etype not in df[n_channel][cell]:
df[n_channel][cell][etype] = {}
if rid not in df[n_channel][cell][etype]:
df[n_channel][cell][etype][rid] = {}
if ch_ not in df[n_channel][cell][etype][rid]:
df[n_channel][cell][etype][rid][ch_] = {}
frame = img.get_image_data("YX", C=idy)
bag = []
bbag = []
for idm, roi in enumerate(rois):
values = get_values(frame, roi)
if roi.roitype==1:
bag.extend(values)
elif roi.roitype==2:
bbag.extend(values)
df[n_channel][cell][etype][rid][ch_]['intensity'] = bag
df[n_channel][cell][etype][rid][ch_]['background'] = bbag
df[n_channel][cell][etype][rid][ch_]['rois'] = rois
df[n_channel][cell][etype][rid][ch_]['img'] = frame
df[n_channel][cell][etype][rid][ch_]['fp'] = fp
df[n_channel][cell][etype][rid][ch_]['pixelsize'] = pixelsize为了便于可视化检查,那么从原始数据开始收集和提取的信息就要尽可能保持完整,而且要遵循一定的逻辑,如上述代码所示,我主要使用字典的方式来装载包含了不同颜色数量,不同细胞,不同实验组,重复组次数,以及成像通道的数据。然后每个数据是xy的frame,并且提取指定ROI(细胞膜区域)的像素,还保留了相关的各种信息,例如文件路径,像素尺寸,roi记录等等,都是为了方便可视化检查。
import matplotlib as mpl
import matplotlib.colors as mcolors
mpl.rcParams['font.family'] = 'DengXian'
plt.rcParams['axes.unicode_minus'] = False
def create_basic_colormap(color_name, num_colors=256):
# 定义黄色范围的RGB值
bcmap = {
'red': (1, 0, 0),
'green': (0, 1, 0),
'blue': (0, 0, 1),
'yellow': (1, 1, 0),
'cyan': (0, 1, 1),
'purple': (1, 0, 1),
'gray': (1, 1, 1)
}
assert color_name in bcmap
colors = []
color = bcmap[color_name]
for i in range(num_colors):
# 计算亮度值 (从0到1)
brightness = i / (num_colors - 1)
# 计算RGB值,注意保持色调,改变亮度
r = color[0] * brightness
g = color[1] * brightness
b = color[2] * brightness
colors.append((r, g, b))
# 创建一个线性分段colormap
cmap = mcolors.LinearSegmentedColormap.from_list(f"{color_name}_gradient", colors)
return cmap
channel_alt = {
'ESID-T1': "BF",
'ESID-T2': "BF",
'AF488-T1': "488",
"AF488-T2": "488",
"AF594-T2": "568",
"AF568-T1": "568",
"AF610-T3": "647",
"AF647-T3": "647",
}
luts = {
'BF': create_basic_colormap('gray'),
'488': create_basic_colormap('green'),
'568': create_basic_colormap('yellow'),
'647': create_basic_colormap('red'),
}
def analyzeSNR(df, n_plex, cell_type, channel, replicate, vmin=0, vmax=2000):
'''计算SNR的同时,显示图像和ROI
n_plex = '2'
cell_type = '97h'
channel = '488'
replicate = 2
vmin = 0
vmax = 2000
'''
cmap = luts[channel]
a = df[n_plex][cell_type]['实验组'][replicate]
b = df[n_plex][cell_type]['mock'][replicate]
res_a = calculateSNR(a[channel]['intensity'], a[channel]['background'])
res_b = calculateSNR(b[channel]['intensity'], b[channel]['background'])
print(f"实验组 SNR: {res_a['SNR']:.2f}, Mock组 SNR: {res_b['SNR']:.2f}")
fig, ax = plt.subplots(ncols=2, nrows=2, figsize=(12,12))
ax[0,0].imshow(a[channel]['img'], cmap=luts[channel], vmin=vmin, vmax=vmax)
ax[0,0].set_title("Exp"+f"\npixelsize: {a[channel]['pixelsize']:.3f} μm\n"+a[channel]['fp'])
ax[1,0].imshow(a['BF']['img'], cmap=luts['BF'])
rois = a[channel]['rois']
for roi in rois:
if roi.roitype==1:
cc = 'red'
elif roi.roitype==2:
cc = 'blue'
pts = roi.coordinates()
ax[0,0].fill(pts[:,0], pts[:,1], facecolor='none', edgecolor=cc, linewidth=1)
ax[1,0].fill(pts[:,0], pts[:,1], facecolor='none', edgecolor=cc, linewidth=1)
ax[0,1].imshow(b[channel]['img'], cmap=cmap, vmin=vmin, vmax=vmax)
ax[0,1].set_title("Mock"+f"\npixelsize: {b[channel]['pixelsize']:.3f} μm\n"+b[channel]['fp'])
ax[1,1].imshow(b['BF']['img'], cmap=luts['BF'])
rois = b[channel]['rois']
for roi in rois:
if roi.roitype==1:
cc = 'red'
elif roi.roitype==2:
cc = 'blue'
pts = roi.coordinates()
ax[0,1].fill(pts[:,0], pts[:,1], facecolor='none', edgecolor=cc, linewidth=1)
ax[1,1].fill(pts[:,0], pts[:,1], facecolor='none', edgecolor=cc, linewidth=1)
plt.tight_layout()
plt.show()
fig, ax = plt.subplots(ncols=2, nrows=1, figsize=(12,3))
ax = ax.ravel()
ax[0].hist(a[channel]["intensity"], alpha=0.5, fc='red', ec='white', label='intensity', bins=30)
ax[0].hist(a[channel]["background"], alpha=0.5, fc='blue', ec='white', label='background', bins=30)
ax[0].set_xlabel("Pixel Value")
ax[0].set_ylabel("Pixel Count")
ax[0].legend()
ax[1].hist(b[channel]["intensity"], alpha=0.5, fc='red', ec='white', label='intensity', bins=30)
ax[1].hist(b[channel]["background"], alpha=0.5, fc='blue', ec='white', label='background', bins=30)
ax[1].set_xlabel("Pixel Value")
ax[1].set_ylabel("Pixel Count")
ax[1].legend()
plt.tight_layout()
plt.show()
res = {
'实验组': res_a['SNR'],
'mock': res_b['SNR']
}
return res可视化检查分析的代码就会变得非常复杂。如上述代码所示,为了使用合适伪彩显示细胞图像,还得先自定义一段代码(后面可以封装起来,作为自己常用的代码模块进行 import);然后用户原始数据中channel name 比较乱,经确认是同一个channel,所以还写了一个 channel_alt 的字典方便做规范化,以便后面批量处理。同时再通过一个 luts 的字典,方便自动给不同的 channel 上合适的伪彩。
由于数据一致性无法保证,所以在 analyzeSNR 函数中,我只能是对每张图自身的信噪比进行分析,这种可以称之为 local SNR。然后每次 analyzeSNR,则会对指定条件和细胞的实验组和对照组数据分别计算 local SNR,然后显示细胞指定通道荧光图像和明场图像,sig 和 bk的ROI区域,原始文件路径,pixelsize等等信息,方便检查。
n_plex = '2'
cell = '97h'
channel = '488'
replicate = 1
print(f"====== {n_plex}-plex, cell: {cell}, channel: {channel}, replicate: {replicate} ======")
res = analyzeSNR(df, n_plex, cell, channel, replicate, vmin=0, vmax=5000)封装好函数之后,具体的调用如上述代码所示,结果如下图:
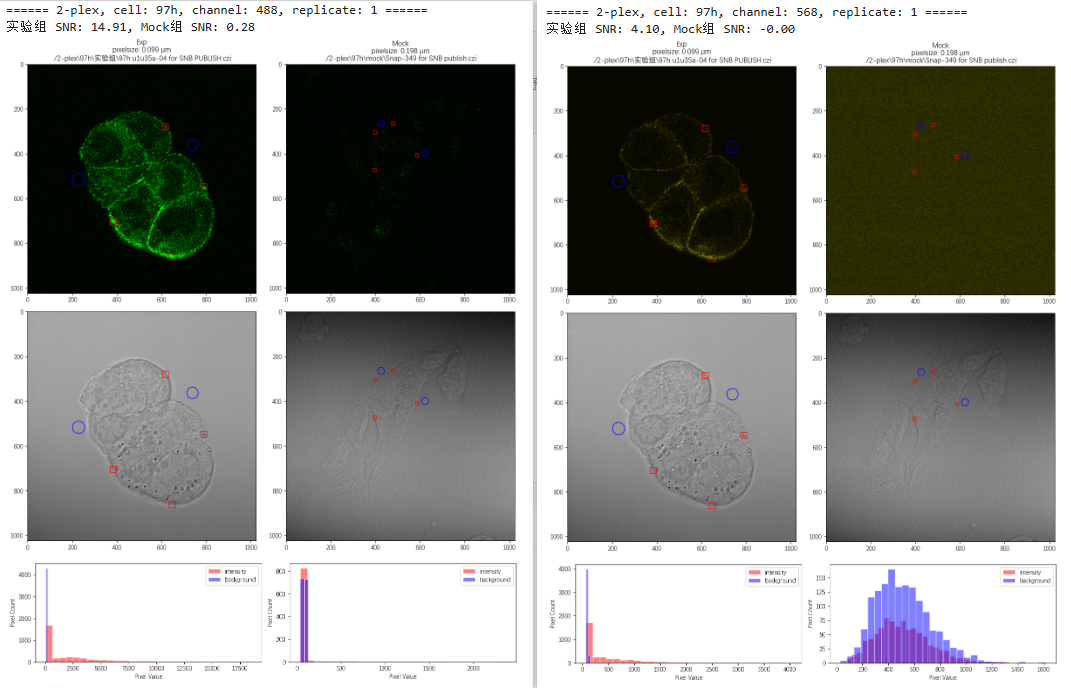
总之,数据可视化是保证数据分析结果准确性的必要检查手段。

此处评论已关闭