这里介绍如何使用cellpose批量分割大量图像,并以此为例展示如何阅读和使用他人的代码。
cellpose提供的用户界面中操作,貌似现在还只能一次处理一张图像。如果需要处理的图像总数量不超过100张,这个也勉强能接受。但如果是成千上万张图像,可能要重复打开文件,点击按钮,保存结果这几个操作很多遍,这个时候就有批量处理的必要了。
cellpose批量处理图像的方法在其源代码仓库中有很完善的示例,而且是 jupyter notebook 格式(后缀为 .ipynb )。相信通过前面的「python基础教程」,我们对这个代码笔记已经很熟悉了。
代码阅读学习

可以直接把cellpose当前的所有源码打包下载到本地(如果下载不便,此文附录提供了百度网盘资源备份)。下载之后解压,然后使用 vscode 打开其中的 notebooks 子目录,界面如下图所示,我们直接打开 run_cyto3.ipynb 这份代码笔记查看。

这份代码笔记内容非常完整,但因为篇幅比较长而且跟常见的文本文献很不一样,所以很容易让人失去阅读的兴趣。事实上对于代码的阅读查看与学习,还是有一定的技巧:
- 搞清楚自己要寻找的核心函数命令
- 搞清楚这个函数的输入输出要求
- 搞清楚这个函数的依赖项
查看图像分割核心命令
在这个例子中,我们想知道的就是怎么调用 cyto3 模型对输入的图像进行分割并输出结果。就按照上述的顺序,快速来寻找需要的关键信息。

打开 vscode 中代码笔记的大纲,可以快速定位到核心的 run segmentation 部分内容。如上图所示,这里首先是实例化了一个对象 model。并且指定了启用 gpu 加速,指定了使用的模型权重为 cyto3,然后调用这个 model 进行处理,就是使用的 model.eval() 方法,输出的有四个东西,那么主要就是下面两行命令:
model = models.Cellpose(gpu=True, model_type="cyto3")
masks_pred, flows, styles, diams = model.eval(imgs,
diameter=0,
channels=[1,2],
niter=2000) 可以看到这个示例中给了四个输入参数:
- imgs:这个就是图像,这个最关键,稍后看看其数据类型要求。
- diameter:这个就类似我们在用户界面上看到的那个 diameter参数,可能需要指定。
- channels:这个在代码注释中有非常多的说明,由于我们要输入的细胞明场图像,不需要处理细胞核,所以设置为
[0, 0]。 - niter:代码注释中说,如果是细菌样品,则需要把这个迭代次数设置高一点。
# define CHANNELS to run segementation on
# grayscale=0, R=1, G=2, B=3
# channels = [cytoplasm, nucleus]
# if NUCLEUS channel does not exist, set the second channel to 0
# channels = [0,0]
# IF ALL YOUR IMAGES ARE THE SAME TYPE, you can give a list with 2 elements
# channels = [0,0] # IF YOU HAVE GRAYSCALE
# channels = [2,3] # IF YOU HAVE G=cytoplasm and B=nucleus
# channels = [2,1] # IF YOU HAVE G=cytoplasm and R=nucleus
# OR if you have different types of channels in each image
# channels = [[2,3], [0,0], [0,0]]
# FOR A CUSTOM MODEL OR OTHER BUILT-IN MODEL:
# model = models.CellposeModel(gpu=True, model_type="livecell_cp3")查看输入数据要求
点击代码大纲中的 images,快速定位到 images 这部分内容,可以看到 imgs 这个变量是如何赋值的。

我们可以尝试运行这段代码,先激活安装了 cellpose 的虚拟环境,vscode 右上角可以选择。然后进入到这个部分的代码块,按 Ctrl+Enter 执行当前代码块,这个时候会下载数据并对 imgs 赋值。

完成赋值之后,我们可以查看这个 imgs 变量的情况:
- 使用
type()函数查看 imgs 的变量类型,发现是 list - 然后取这个 list 中第一个 item 进一步查看,发现是 numpy 的矩阵对象
- 接着查看这个矩阵的数值类型,是 float32,但是我估计输入图像是 8-bit(uint8) 也没关系
- 再查看这个矩阵的shape,是
(2, 383, 512)对应(channels, rows, cols)这几个维度,说明这个图像包含两个通道,高度为383,宽度为512像素。 - 使用简单的 matplotlib 作图查看这个数据。

执行图像分割并调参尝试
前面已经试着下载并查看了数据,要想查看输出的结果,就要执行图像分割。所以我们先回到 run segmentation 部分执行一下,会出现如下报错:
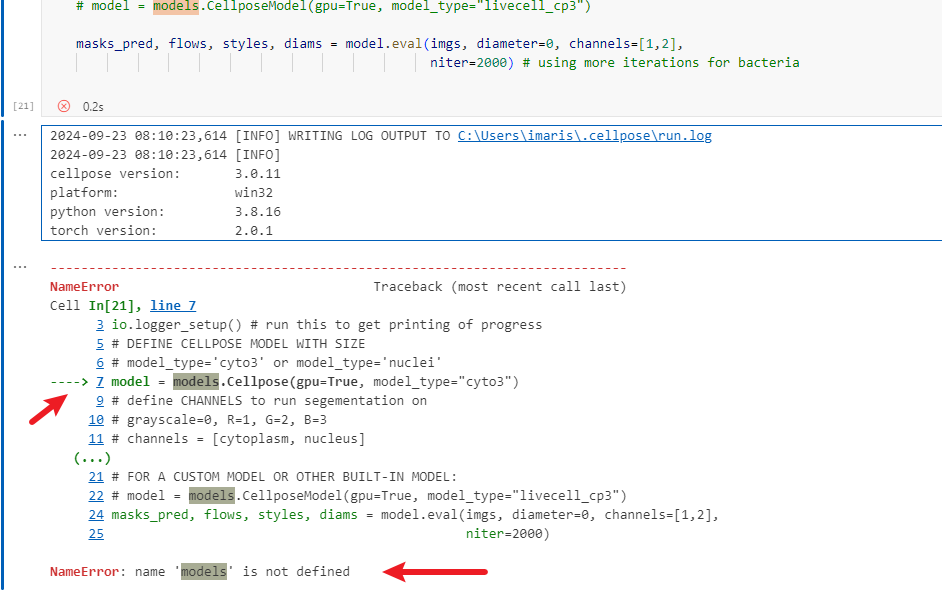
python出错时,命令行中出会出现一个错误堆栈。最后的Error小结是 models 这个变量没有定义,然后从下往上看,可以看到是第7行代码出错。这个 models 要么是前面的代码中有 define,要么是从别的 module 中 import,不管是哪种情况,我们可以在当前代码笔记中搜索 models。

很快就在最开始的 block 中找到了 models 的定义语句,它是从 cellpose 模块 中 import 进来的。而这个也是我们后续使用中必须要进行的步骤,以确保核心命令执行所需的依赖项完整。这里我们就执行下这个部分的代码。

可以看到这里的运行日志输出,首先是对 cell 的 diamter 进行了估计,耗时 14 秒左右。这提示 diameter 参数设置为0的时候会自动估计(相对耗时)。然后 find mask 得速度就很快了,24张图像就用了 13 秒搞定。
接下来我们可以尝试只对其中一张图像的一个通道进行分割,并指定 diameter,修改 channels以指向为明场类型。

查看图像分割结果
上部分我们调整了输入和参数来执行图像分割,output 为 masks_pred 等四个变量。不难猜测,我们需要的 mask 图像就放在 masks_pred 这个变量中。可以查看一下:

所以 imgs[0][0] 指向的这张单通道的图像(xy类型)predict 之后对应的mask就是 masks_pred[0],概括下就是 input 和 output 的两个 list 的item 的index 是一致的。
代码消化整合
基于上述代码阅读获得的必要信息,我们可以新建一个notebook,进行尝试。
对图像分割结果作图如下所示:

可以看到,最后一张图像啥也没分割出来,这可能是自动设置的 diameter 不合适。所以说,针对同一类型的细胞,所有图像的 pixelsize 要调整一致,然后大致测量下,提供一个估计的 diameter 再来进行 cellpose 的图像分割。
附录
通过百度网盘分享的文件:cellpose_cyto3_notebooks.zip
链接:https://pan.baidu.com/s/1Sls8hzZQsUfduFAGnJ5eqg
提取码:

此处评论已关闭